
 Article
Article
 04.03.2025
04.03.2025
Перевага цилостазолу серед антитромбоцитарних препаратів, затверджених FDA, щодо можливого впливу на COVID 19. (Mpro та спайковий білок). Перепрофілювання ліків
Мухаммед А. Абошеаша 1 | Афнан Х. Ель-Говілі 2,3; 1) Лабораторія клітинної генетики, Токійський столичний університет, Токіо, Японія; 2) Кафедра хімії, Відділ біохімії, Природничий факультет, Університет Танта, Танта, Єгипет; 3) Кафедра фізіології органів і клітин, Університет Джунтендо, Токіо, Японія
Superiority of cilostazol among antiplatelet FDA-approved drugs against COVID 19 Mpro and spike protein: Drug repurposing approach
Abstract
Coronavirus disease 2019 (COVID 19) was first identified in Wuhan, China near the end of 2019. To date, COVID-19 had spread to almost 235 countries and territories due to its highly infectious nature. Moreover, there is no vaccine or Food and Drug Administration (FDA)-approved drug. More time is needed to establish one of them. Consequently, the drug repurposing approach seems to be the most attractive and quick solution to accommodate this crisis. In this regard, we performed molecular docking-based virtual screening of antiplatelet FDA-approved drugs on the key two viral target proteins: main protease (Mpro) and spike glycoprotein (S) as potential inhibitor candidates for COVID-19. In the present study, 15 antiplatelet FDAapproved drugs were investigated against the concerned targets using the Molecular Docking Server. Our study revealed that only cilostazol has the most favorable binding interaction on Mpro (PDB ID: 6LU7) and cilostazol, iloprost, epoprostenol, prasugrel, and icosapent ethyl have a higher binding affinity on spike glycoprotein (S) (PDB ID: 6VYB) compared with recent anti-CoVID-19. Therefore, cilostazol is a promising FDA drug against COVID-19 by inhibiting both Mpro and S protein. The insights gained in this study may be useful for quick approach against COVID-19 in the future.
KEYWORDS
antiplatelet, COVID-19, molecular docking, Mpro, SARS-CoV-2, spike glycoprotein
1 | В СТУП
Всесвітньою організацією охорони здоров’я (ВООЗ). Станом на 11 березня 2020 р. ВООЗ заявила, що COVID ‐ 19 віднесено до категорії пандемії (Ramphul & Mejias, 2020 ). До 23 квітня 2020 року було зафіксовано 2 397 217 підтверджених випадків та 162 956 смертей у 235 країнах та територіях [Всесвітня організація охорони здоров’я (ВООЗ), 2020 ]. SARS-CoV-2 – це оболонка позитивного одноланцюгового РНК-вірусу, класифікованого з сімейства бетакоронавірусів (β-CoV), який містить інших представників, включаючи SARS-CoV та MERSCoV (Chan et al., 2015 ; Salata, Calistri, Parolin, & Palù, 2019 ). Вірусний нуклеокапсид складається з РНК геномного гена і нуклеокапсидного білку (N), який вбудований всередину фосфоліпідних бішарів і захищений двома окремими формами колосових білків: шиповим тримером глікопротеїну (S) у всіх CoV і гемаглютинін-естеразою HE в деяких CoVs. Мембранний білок (М) (трансмембранний глікопротеїн III типу) та оболонковий білок (Е) розміщені серед S-білків у вірусній оболонці. CoV були названі на основі зовнішнього вигляду у формі корони (Li et al., 2020). Орієнтовна структура ГРВІ-CoV-2 наведена на рисунку 1.
.jpg)
РИСУНОК 1 Орієнтовна структура ГРВІ-CoV-2
SARS-CoV-2 викликає важку інфекцію дихальних шляхів у людей, використовуючи рецептори ангіотензинперетворюючого ферменту 2 (ACE2) як ворота для зараження епітеліальних клітин легенів шляхом приєднання шипового глікопротеїну (S) (Chen, Guo, Pan, & Zhao, 2020 ). Геномну послідовність ГРВІ-CoV-2 було виділено та отримано Lu et al. (2020). Також кристалічну структуру основної протеази COVID-19 (Mpro) підтвердили Jin et al. що розглядають її як потенційний цільовий білок для інгібування реплікації ГРВІ-CoV-2. Mpro є ключовим білком у запобіганні дозріванню вірусу (Jin et al., 2020 ). Отже, націлювання на неструктурні (Mpro) та структурні( S) білки має перспективний підхід для ефективного лікування проти ГРВІ-CoV-2 (Sohag et al., 2020 ).
Вчені досліджують альтернативні методи лікування COVID-19, використовуючи штучний інтелект. Багато дослідників, що працюють у галузі переназначення ліків, використовують Банк препаратів та програмне забезпечення для молекулярного стикування, щоб сподіватися знайти потенційне лікування. Потенційне призначення лікарських засобів (також його часто називають репозиціонуванням лікарських засобів) – це стратегія розробки лікарських засобів, яка використовується для виявлення нових застосувань існуючих схвалених та досліджуваних лікарських засобів поза їх початковими показаннями. У порівнянні зі звичайними можливостями для виробництва ліків, цей підхід має багато переваг. На відміну від звичайного виробництва лікарських засобів, яке може бути неефективним у доклінічних та ранніх стадіях клінічних випробувань, заснованих на питаннях безпеки, цей ризик пом’якшується використанням препаратів, які продемонстрували дані безпеки в попередніх дослідженнях. У минулих пандеміях респіраторного вірусу, таких як грип H1N1, використовували терапевтичні антикоагулянти (Obi et al., 2019). Нещодавнє дослідження свідчить, що використання гепарину як профілактичного засобу у 99 пацієнтів було пов’язане із зменшенням смертності в когортному дослідженні 449 пацієнтів з COVID-19 з Уханю, Китай. Однак рівень профілактичних антикоагулянтів був низьким, для підтвердження цієї гіпотези необхідні подальші проспективні дослідження (Tremblay et al., 2020). Крім того, лікарня у Xijing розпочала клінічне випробування, пропонуючи раннє використання аспірину. Це, як очікується, зменшить частоту важких та критичних хворих на COVID-19, мінімізує перебування в лікарні та попередить виникнення серцево-судинних ускладнень на основі ролі аспірину, щодо реплікації вірусу, агрегації тромбоцитів, розвитку інфекції та пошкодження легень (NCT04365309, 2020); що ставить питання про те, чи можуть тромбоцити відігравати роль у лікуванні COVID ‐ 19.
2 | МАТЕРІАЛ І МЕТОДИ
2.1 | Молекулярна док-платформа
Обчислювальні дослідження проводились із використанням Molecular Docking Server (Bikadi & Hazai, 2009 ) (https://www.dockingserver.com ) на основі AutoDock 4 для розрахунку стикування. Часткові заряди білка та ліганду були розраховані методом PM6 із використанням програмного забезпечення MOPAC2009 (Huey, Morris, Olson, & Goodsell, 2007 ; Stewart, 2009 ).
2.2 | Визначення та підготовка ліганду
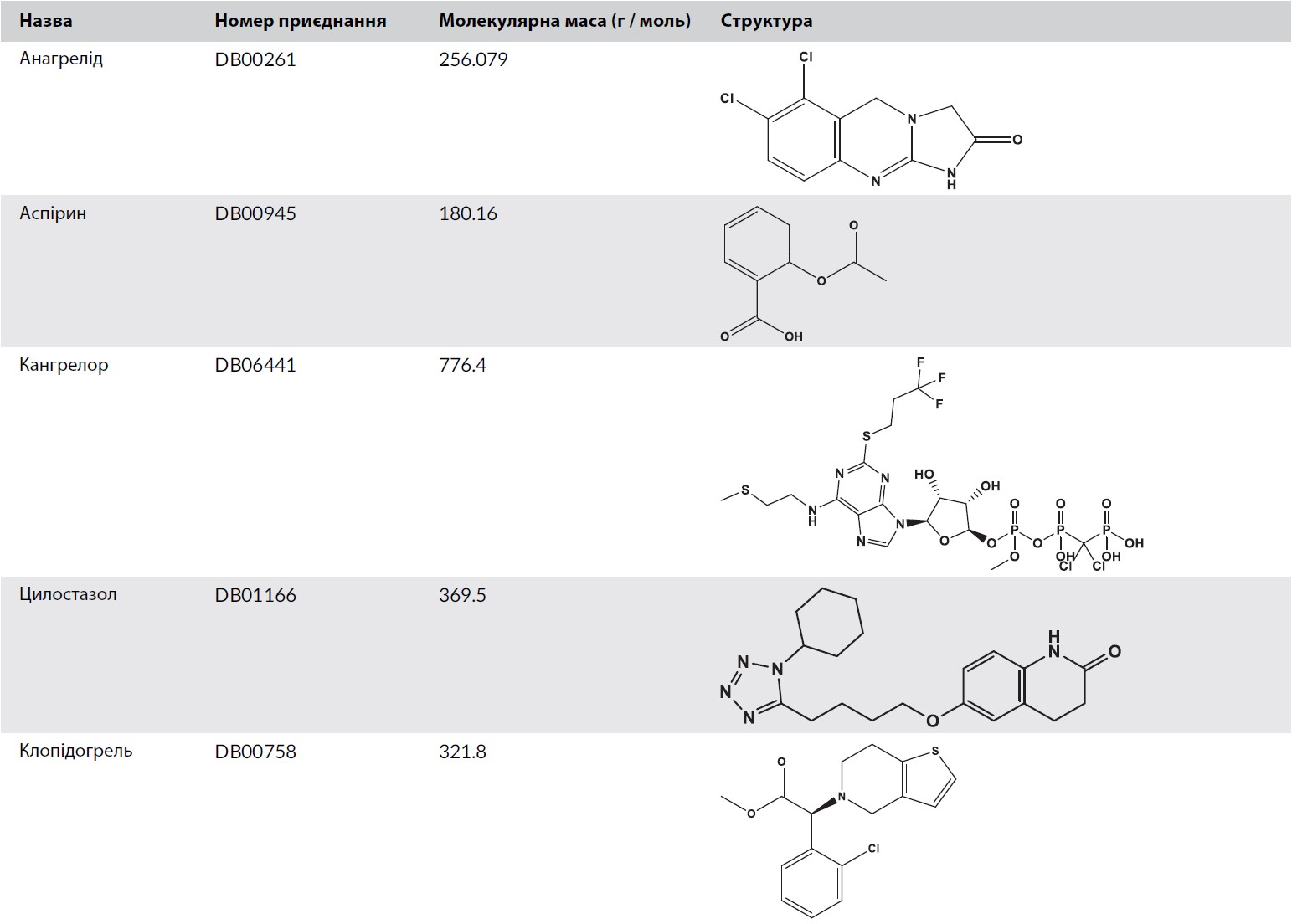
Згідно з базою даних Drugbank, було обрано 47 антиагрегантних препаратів (DBCAT000149), агентів, які протидіють будь-якому механізму, що призводить до агрегації тромбоцитів крові. З них було вибрано 15 відносно нових молекул, що схвалені FDA (таблиця 1) та порівняно з тими що вже використовувються та є передбачуваними інгібіторами COVID-19, наведеними в таблиці 2 . База даних PubChem була використана для вилучення тривимірних (3D) хімічних структур вибраних молекул. 3D-оптимізацію та оптимізацію геометрії з мінімізацією енергії лігандів виконували за допомогою алгоритмів, що контролюються на Docking Server. Модуль підготовки лігандів використовував включене «Поле молекулярних сил Merck 94» (MMFF94), як метод оптимізації геометрії та « Gasteiger» , як метод розрахунку заряду при рН 7.
ТАБЛИЦЯ 1. Список антитромбоцитарних препаратів, схвалених FDA, щодо можливого впливу на COVID-19
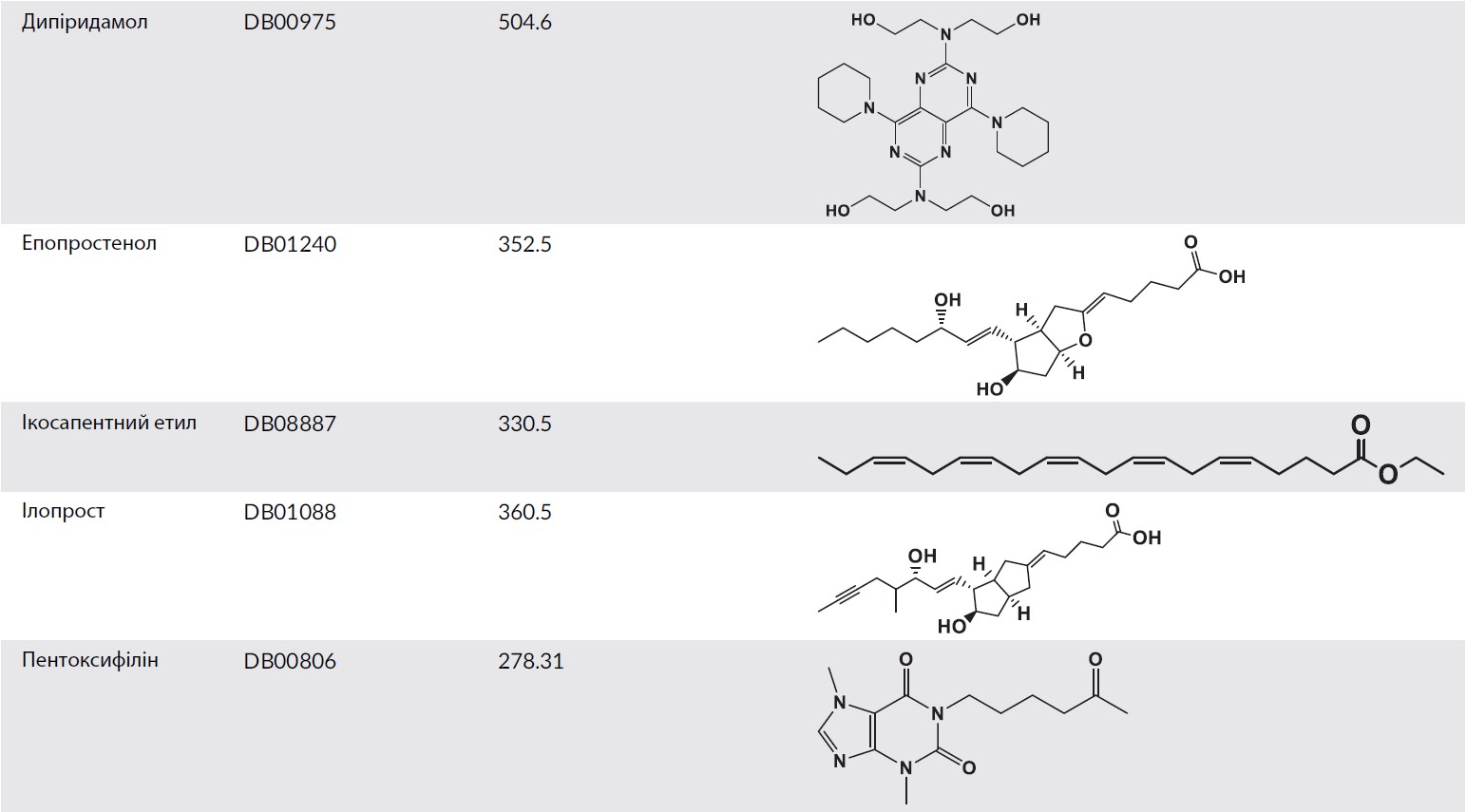
ТАБЛИЦЯ 2. Список антитромбоцитарних препаратів, схвалених FDA, щодо можливого впливу на COVID-19
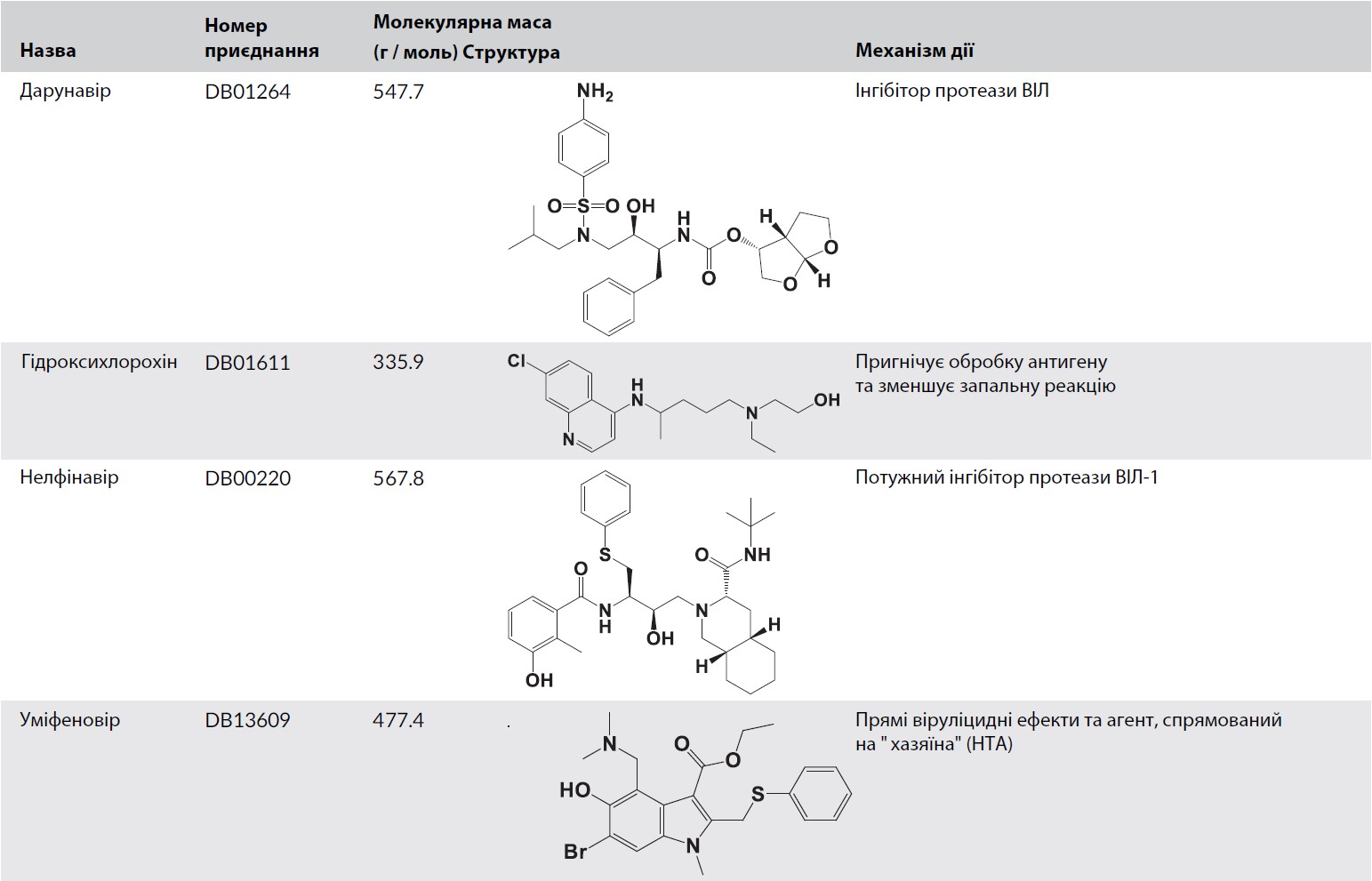
2.3 | Визначення та підготовка білка
В якості мішеней інгібування для лікарських засобів були обрані два білки SARS-CoV-2: основна протеаза (Mpro) (PDB ID: 6LU7) (Jin et al., 2020 ) та гликопротеїн із шипами (S) (PDB ID: 6VYB) (Walls et al. ., 2020 ), отримані з Банку даних білків RCSB ( http://www.rscb.org ). Білкові структури були підготовлені за допомогою майстра підготовки білка на панелі Molecular Docking Server. Були присвоєні порядки зв’язку та додані атоми водню. Видалено молекули води та інші неспецифічні молекули. Карти спорідненості (сітки) точок сітки 20 × 20 × 20 Å та інтервал 0,375 Å були сформовані за допомогою програми Autogrid (Morris et al., 1998). Набір параметрів AutoDock та залежні від відстані діелектричні функції використовувались при розрахунку ван-дер-Ваальса та електростатичних даних, відповідно.
2.4 | Обчислювальні методи
Моделювання стикування роводили з використанням генетичного алгоритму Ламарккі (LGA) та методу локального пошуку Соліса та Ветса (Solis & Wets, 1981). Початкове положення, орієнтація та кручення молекул ліганду були встановлені випадковим чином. Кожен експеримент з док-станцією був проведений на основі 100 різних прогонів, які було завершено після максимум 2 500 000 оцінок енергії. Чисельність популяції була встановлена на 150. Під час пошуку застосовували поступальний крок 0,2 Å, а кватерніон і кручення 5. Після кожного розрахунку стикування оцінювали середньоквадратичне відхилення (RMSD) між даними ліганду з найменшою енергією та даними ліганду складної кристалічної структури. Вибір стану, даних з найнижчим RMSD визначався з усіх даних, отриманих програмою стикування. Вихідні результати сервера молекулярного докінгу оцінювали вільну енергію зв’язування (ккал / моль) як Δ Gзначення. Далі розраховували константи гальмування ( K i ). Значення K i для аналізованих даних стикування розраховували за параметрами Δ G наступним чином (Huey et al., 2007):
.jpg)
де, R (газова константа) дорівнює 1.98 кал (моль K)−1, а T (кімнатна температура) – 298,15 K.
Після стикування комплекси аналізували за допомогою веб-сервера Protein-Ligand Interaction Profiler (PLIP) (Технічний університет Дрездена) (Salentin, Schreiber, Haupt, Adasme та Schroeder, 2015).
3 | РЕЗУЛЬТАТИ ТА ОБГОВОРЕННЯ
У поточному дослідженні параметри розрахункової вільної енергії зв’язування, константи гальмування ( K i ), загальної розрахункової енергії vdW + Hbond + дезольв (EVHD), електростатичної енергії, загальної міжмолекулярної енергії, частоти зв’язування та площі взаємодіючої поверхні були оцінені для сприятливого зв’язування антитромбоцитарних препаратів, схвалених FDA, щодо можливого впливу проти COVID‐19 (Mpro) та гликопротеїну з шипами (S).
3.1 | Молекулярне стикування антиагрегантних препаратів, схвалених FDA проти COVID-19 (Mpro)
Результати молекулярного стикування показали, що цилостазол має найбільш сприятливу взаємодію зв’язування на Mpro (PDB ID: 6LU7) з розрахунковою вільною енергією зв’язування -8,48 ккал / моль та константою інгібування (Ki) 612,08 нМ. При застосуванні нелфінавіру з розрахунковою вільною енергією зв’язування -7,69 ккал / моль, а константа інгібування (Ki) 2,31 мкМ. Нещодавно повідомлялося, що гідроксихлорохін, уміфеновір та дарунавір мають потужний інгібуючий ефект проти ГРВІ-CoV-2 (Devaux, Rolain, Colson, & Raoult, 2020; Гаррісон, 2020; Ван, Чень, Лу, Чень, & Чжан, 2020). Вони виявили нижчу спорідненість до зв’язування з Mpro за оцінкою вільної енергії зв’язування -7,06, -6,51 і -6,47 ккал / моль, відповідно, ніж тикагрелор, тиклопідин та прасугрел, з енергією зв’язування -7,51, -7,34 та -7,29 ккал / моль відповідно (Таблиця 3).
ТАБЛИЦЯ 3. Результати стикування антитромбоцитарних препаратів, схвалених FDA, у порівнянні із загальними інгібіторами на кристалічній структурі COVID-19 (Mpro ) (PDB ID: 6LU7)
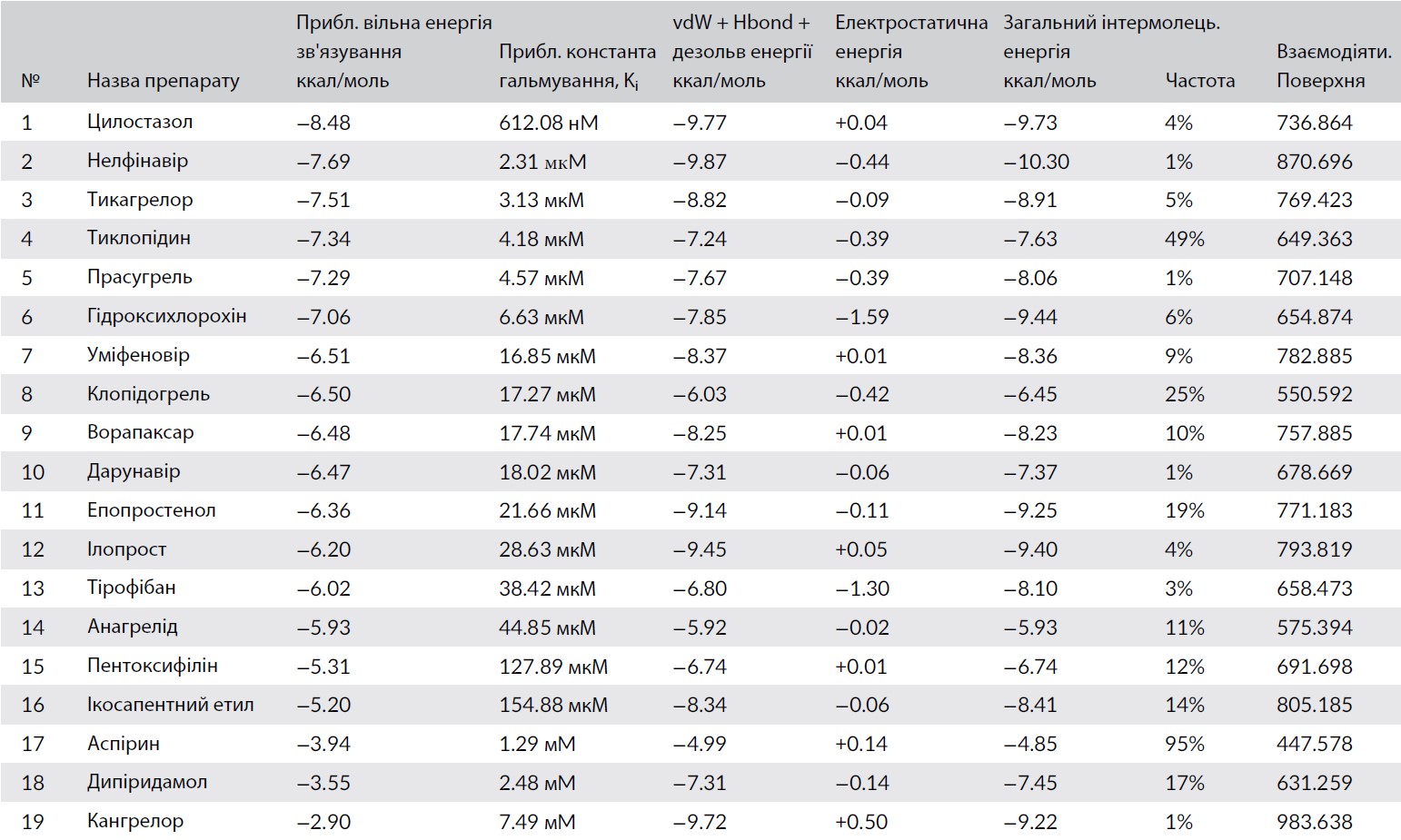
Рисунок 2 демонструє аналіз PLIP для стикованих конструкцій Mpro до цилостазолу (малюнок 2a) та нелфінавіру (малюнок 2b). Mpro показано на кольоровій моделі. Ліганди показані в помаранчевих кольорах. Збільшений вигляд показує, взаємодію при стикуванні. Залишки місця зв’язування Mpro представлені синіми паличками та позначені як трибуквенний код. На рисуноку 2 представлені гідрофобні взаємодії, описані штрихово-сірими лініями, тоді як Н-зв’язки проілюстровані як суцільні сині лінії. Цікаво, що більшість зв’язків переважно негідрофобні, тоді як у всіх загальних стикувальних комплексах переважають гідрофобні взаємодії.
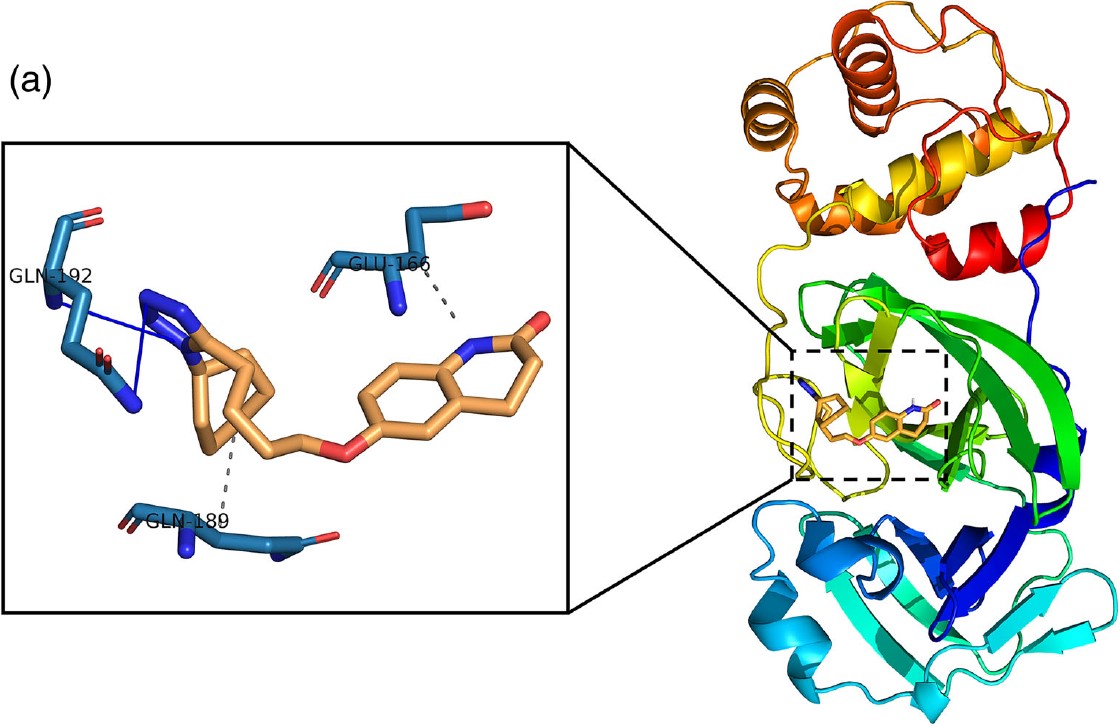
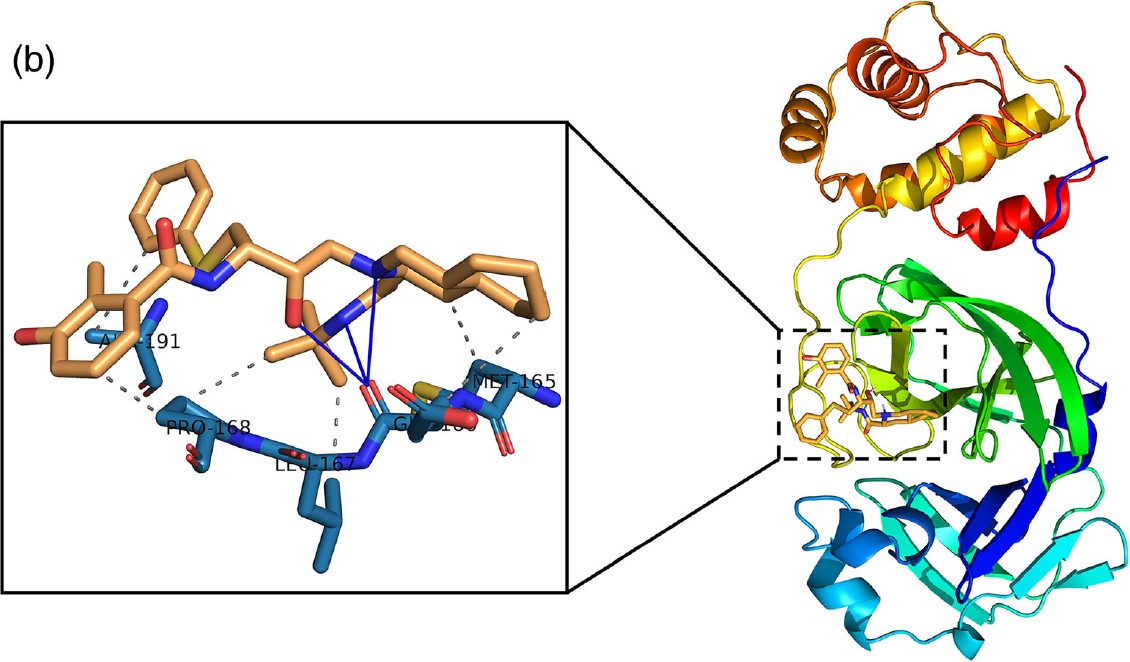
РИСУНОК 2 Прогнозовані моделі зв’язування, отримані в результаті імітаційного аналізу цилостазолу та нелфінавіру проти основної протеази COVID-19 (Mpro). Структура, показана як модель поверхні Mpro. Цилостазол і нелфінавір представлені у вигляді помаранчевої палички.
(а) Комплекс SARS-CoV-2 Mpro– цилостазол.
(b)Комплекс SARS-CoV-2
Mpro – нелфінавір. Залишки активної ділянки в розширених панелях представлені синіми паличками. Н-зв’язки та гідрофобні взаємодії відображаються відповідно синіми пунктирно-штриховими лініями
3. 2 | Молекулярне стикування антитромбоцитарних препаратів затверджених FDA, щодо можливого впливу на глікопротеїн (S) COVID-19
SARS-CoV-2 може потрапляти до клітини-хазяїна, зв’язуючись з ангіотензином людини, використовуючи рецептор ферменту 2 (hACE2) за допомогою спайкового глікопротеїну (S). Тобто націлювання на білок (S) відіграє ключову роль у лікуванні COVID-19 (Ou et al., 2020). Крім того, результати молекулярного стикування антитромбоцитарних препаратів, схвалених FDA, щодо можливого впливу на глікопротеїн COVID-19 із шипами (S) показали, що цилостазол, ілопрост, епопростенол, прасугрель, та ікосапентовий етил мають найбільш перспективну взаємодію з зв’язуванням спайкового глікопротеїну (S) (PDB ID: 6VYB); розрахункова вільна енергія зв’язування −9,97, −9,68, −9,07, −9,00 та −8,29 ккал / моль відповідно, і константа інгібування (Ki) 48,86, 80,24, 222,84, 251,80 і 842,93 нМ, відповідно. Тоді як нелфінавір виявляв нижчу спорідненість до (PDB ID: 6VYB) з розрахунковою вільною енергією зв’язування -7,78 ккал / моль, і константою інгібування (Ki) 1,98 мкМ (таблиця 4).
ТАБЛИЦЯ 4. Результати стикування антитромбоцитарних препаратів, схвалених FDA, порівняно із загальними інгібіторами на кристалічній структурі глікопротеїну COVID-19 із шипами (S) (PDB ID: 6VYB)
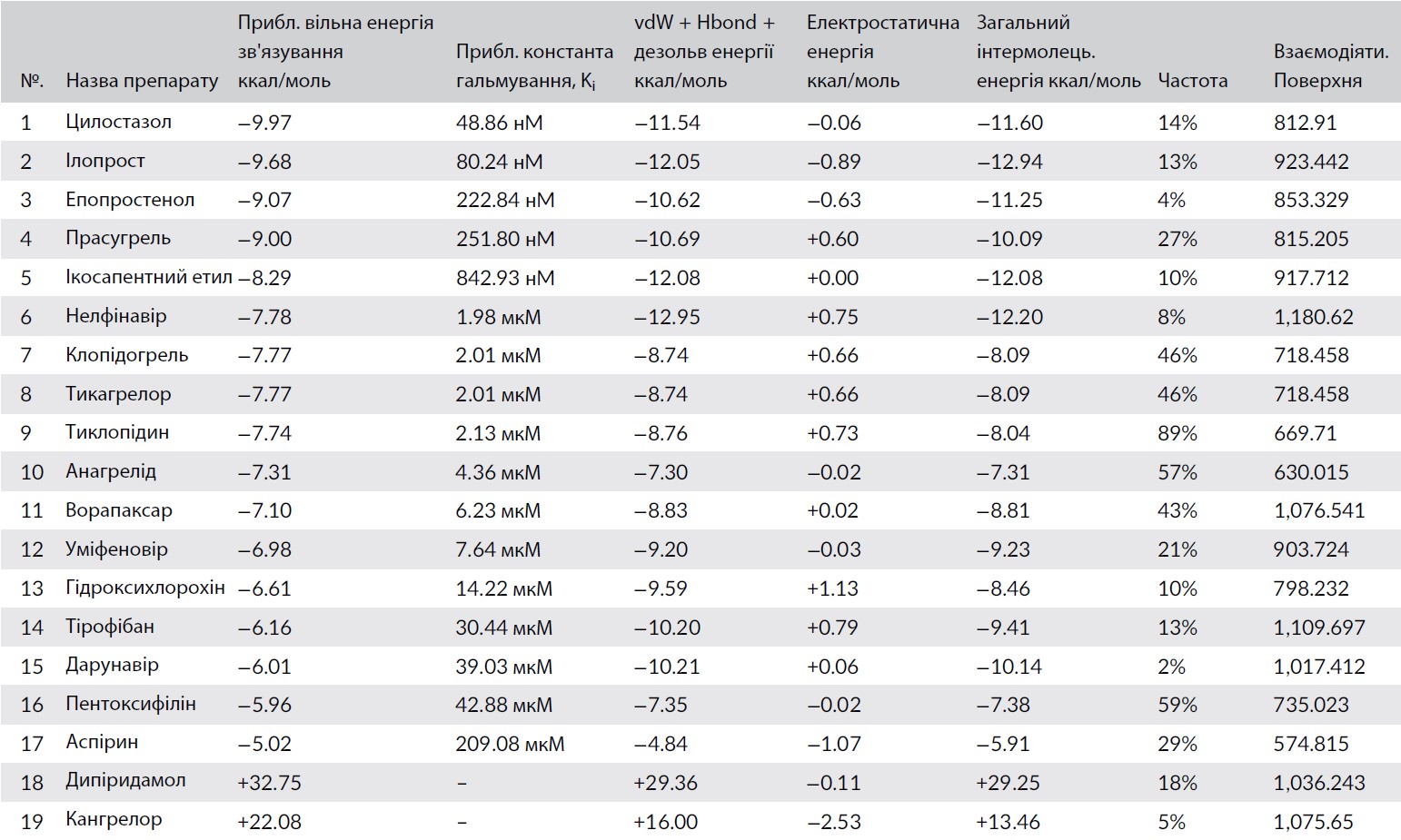
З іншого боку, інші досліджені антитромбоцитарних препаратів, схвалених FDA, таких як: клопідогрель, тикагрелор, тиклопідин, анагрелід та ворапаксар показали кращу спорідненість до зв’язування з розрахунковою вільною енергією зв’язування −7,77, −7,77, −7,74, −7,31 та −7,10 ккал / моль відповідно, ніж уміфеновір, гідроксихлорохін та дарунавір із розрахунковим вмістом вільної енергії зв’язування −6,98, −6,61 та −6,01 ккал / моль відповідно (табл. 4).
Рисунки 3 і 4 ілюструють аналіз PLIP для стикованих структур спайкового глікопротеїну (S) до цилостазолу, ілопросту, епопростенолу, прасугрелю, ікосапенту етилу та нелфінавіру відповідно. Шипований глікопротеїн (S) показаний на кольоровій моделі поверхні стрічки. Ліганди показані в помаранчевих паличках, де зв’язують глікопротеїн (S). Збільшення показує, як встановлюється взаємодія при стикуванні. Місця зв’язування спайкового глікопротеїну продемонстровані синіми паличками та позначені трибуквеним кодом. Гідрофобні взаємодії описані пунктирно-сірими лініями, тоді як Н-зв’язки проілюстровані суцільними синіми лініями. Як показано в Mpro лігандні взаємодії, а також гідрофобні взаємодії є домінуючими в усіх комплексах, що стикуються з білками. Для прасугрелю з Q 1010C будується лише один водневий зв’язок, тоді як у нелфінавірі є сім водневих зв’язків.
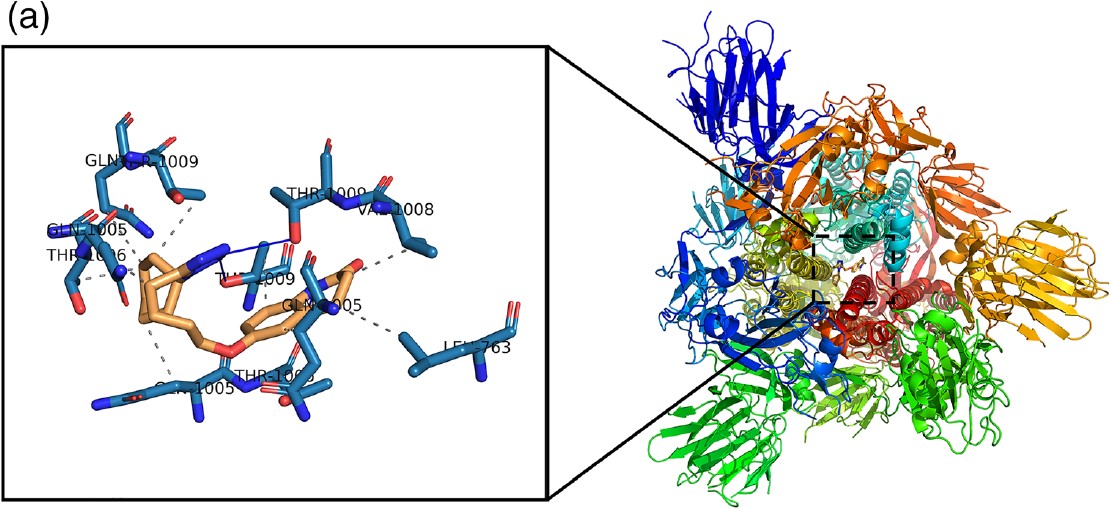
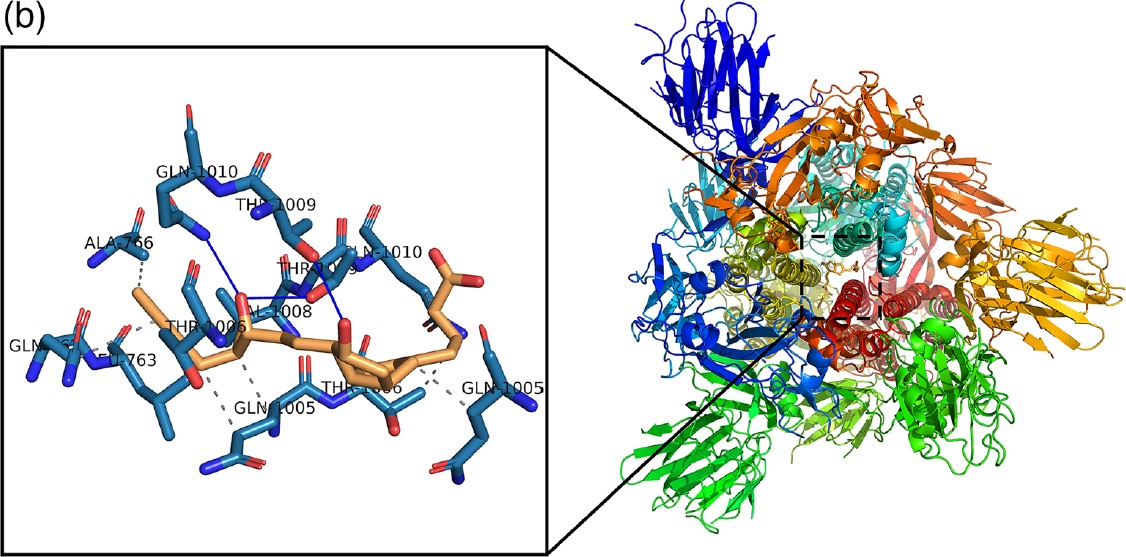
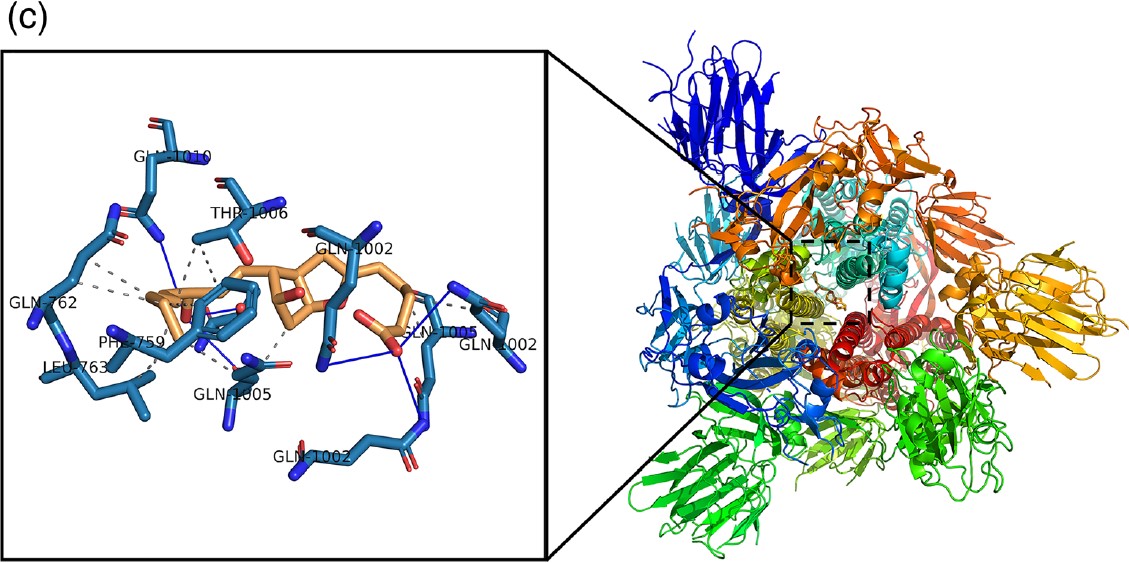
РИСУНОК 3 Прогнозовані моделі зв’язування, отримані в результаті імітаційного аналізу цилостазолу, ілопросту та епопростенолу проти COVID-19 Spike
Glycoprotein (S). Структура (S) білка показана як модель поверхні стрічки. Цилостазол, ілопрост та епопростенол представлені у вигляді помаранчевої палички.
(a) Комплекс SARS-CoV-2 (S) – цилостазол.
(b) SARS-CoV-2 (S) – комплекс ілопросту.
(c) SARS-CoV-2 (S) – епопростенольний комплекс.
Залишки активної ділянки в розширених панелях представлені синіми паличками. Н-зв’язки та гідрофобні взаємодії відображаються відповідно синіми пунктирно-штриховими лініями
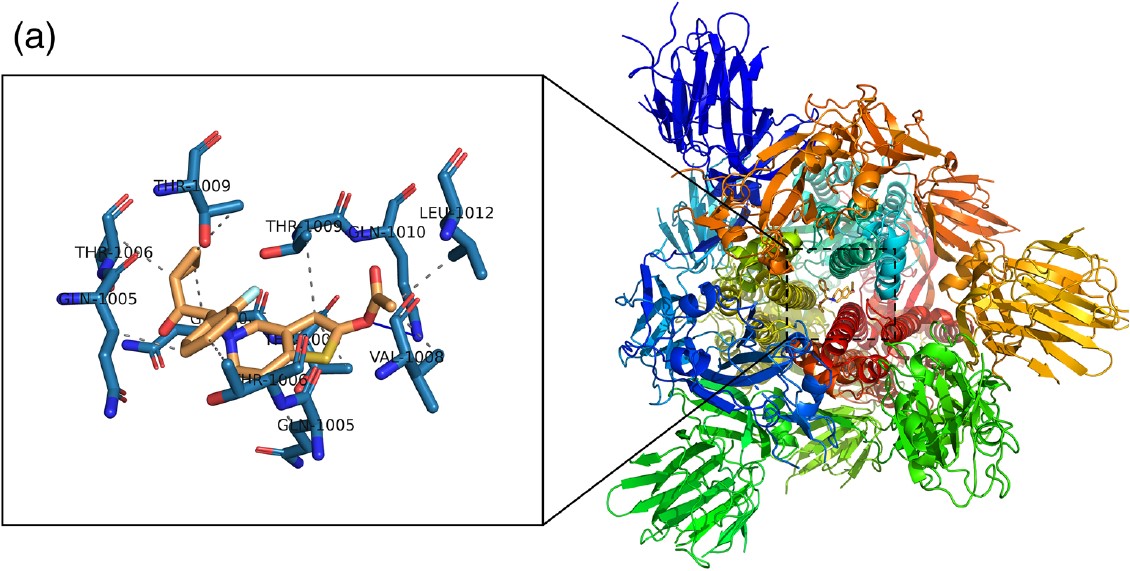
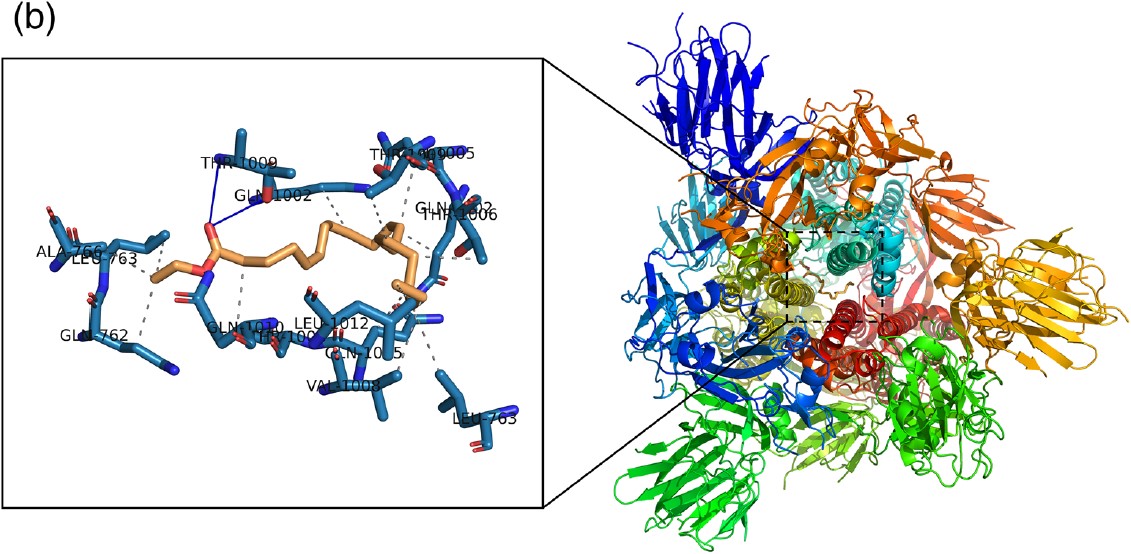
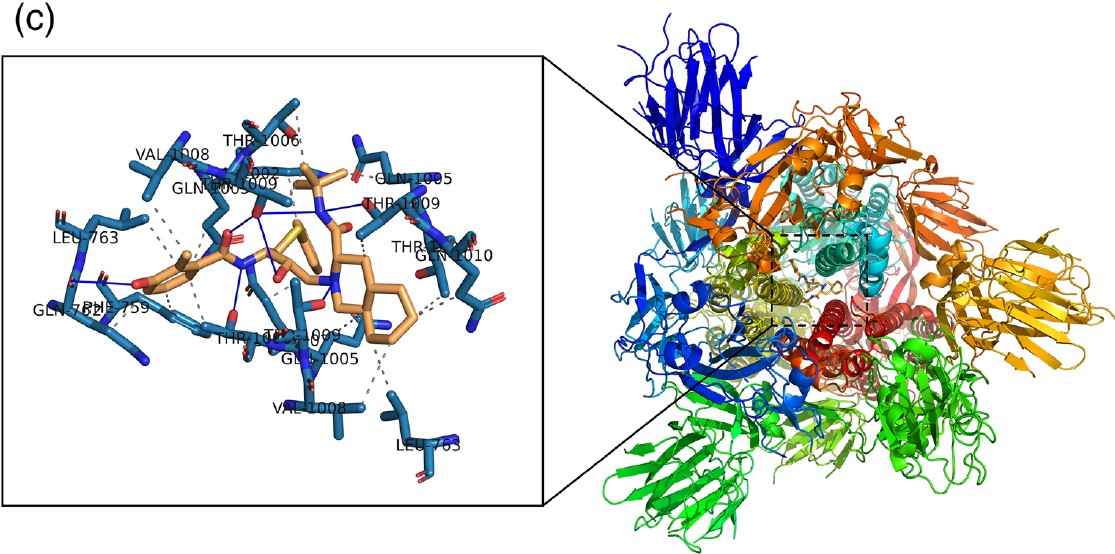
РИСУНОК 4 Прогнозовані моделі зв’язування, отримані в результаті імітаційного аналізу стикувальних речовин прасугрелю, ікосапенту етилу та нелфінавіру проти глікопротеїну COVID-19 із шипом (S). Структура (S) білка показана як модель поверхні стрічки. Прасугрель, ікосапент етил та нелфінавір представлені у вигляді помаранчевої палички.
(a) Комплекс SARS-CoV-2
(S) – прасугрель.
(b) SARS-CoV-2 (S) – ікосапентний етиловий комплекс.
(c) Комплекс SARS-CoV-2 (S) – нельфінавір.
Залишки активної ділянки в розширених панелях представлені синіми паличками. Н-зв’язки та гідрофобні взаємодії відображаються відповідно синіми пунктирно-штриховими лініями
У таблицях 5 та 6 узагальнено взаємодію, встановлену між антитромбоцитарними лікарськими засобами, схваленими FDA, та цільовими білками Mpro та білком S відповідно. Домінуючими є два типи взаємодій – Н-зв’язок та гідрофобні взаємодії. Крім того, повідомляється про галогенний зв’язок (залишки виділені жирним шрифтом у таблицях) між залишками Mpro E 166, Q 189 та F 140 з прасугрелем, клопідогрелем та анагрелідом відповідно або між залишковим білком Q 1002C та анагрелідом. Гідрофобні взаємодії є більш домінуючими порівняно з H-зв’язком, як це видно з майже всіх вибраних антитромбоцитарних препаратів, схвалених FDA, з білком Mpro та S.
ТАБЛИЦЯ 5 Взаємодія, побудована між антитромбоцитарними лікарськими засобами, схваленими FDA, та ГРВІ-CoV-2 Mpro
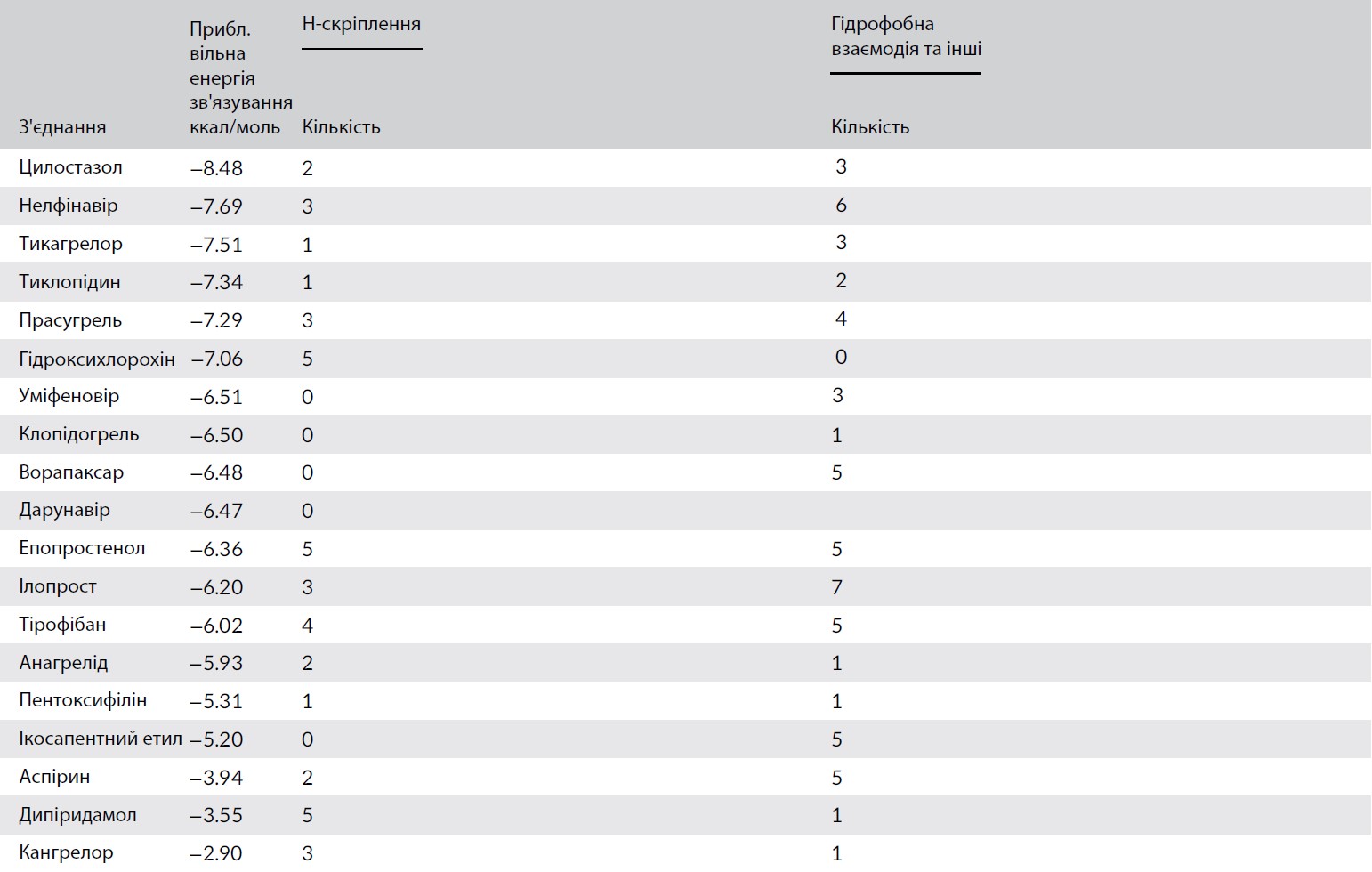
Примітки: Залишки взаємодіють через галогенний зв’язок.
ТАБЛИЦЯ 6. Взаємодія, побудована між антитромбоцитарними лікарськими засобами, схваленими FDA, та SARS ‐ CoV ‐ 2 Spike protein
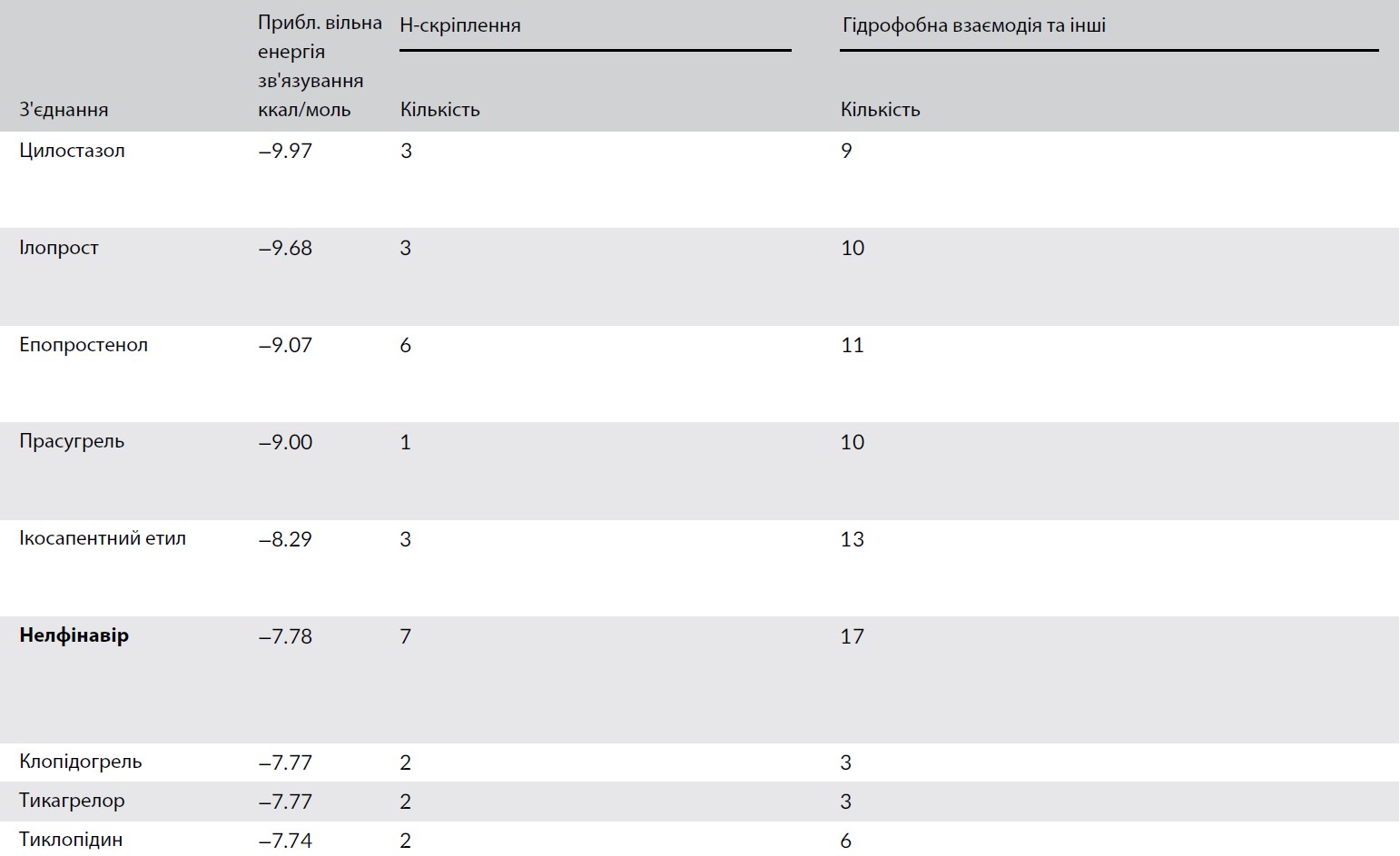
Примітки: Залишки взаємодіють через галогенний зв’язок.
На рисунку 5 узагальнено оцінено енергію вільного зв’язування антитромбоцитарних препаратів, схвалених FDA, щодо можливого впливу на COVID ‐ 19 (Mpro ) та спайкового глікопротеїну (S) і показано, що цилостазол має найнижчу енергію вільного зв’язування −8,48 та −9,97 ккал / моль серед Mpro та шипу глікопротеїну, відповідно, що свідчить про те, що цилостазол є перспективним препаратом для інгібування як Mpro, так і S білка при лікуванні COVID ‐ 19.
.jpg)
РИСУНОК 5 Оціночна вільна енергія зв’язування (-ккал/моль) для антитромбоцитарних препаратів, схвалених FDA з основною протеазою COVID-19 (PDB ID: 6LU7) та білком (PDB ID: 6VYB)
4 | ВИСНОВОК
Протягом останніх кількох місяців спалах СОВІД-19, що швидко поширюється, спричинив виклики для світового ринку охорони здоров’я. На сьогоднішній день не існує ефективної вакцини або затверджених ліків для лікування цієї хвороби. З огляду на час, необхідний для створення однієї з цих альтернатив, підхід до перенаправлення ліків видається найбільш привабливим та швидким. Щоб допомогти протистояти COVID-19, був проведений віртуальний молекулярний скринінг для класифікації антитромбоцитарних препаратів, схвалених FDA, які здатні зв’язуватись COVID-19 з білком Mpro і S. Серед усіх антитромбоцитарних препаратів, схвалених FDA, цилостазол показав найбільшу перспективу проти COVID-19, інгібуючи як Mpro, так і S білок. Для того, щоб перетворити ці потенційні інгібітори на терапевтичні ліки, більше in vitro та in vivoнеобхідні тести. Отримані в цьому дослідженні ідеї можуть бути корисними для вивчення та розробки нових терапевтичних засобів проти COVID-19 у майбутньому.
ЗАЯВА ПРО ДОСТУПНІСТЬ ДАНИХ
Набори даних, створені та / або проаналізовані під час поточного дослідження, є такими можна отриматиу відповідного автора за запитом.
ЕТИЧНА ЗАЯВА
Автори заявляють, що немає конфлікту інтересів щодо фінансових та / або особистих стосунків з іншими людьми чи організаціями, які могли б неналежним чином вплинути на цю роботу. Жодної роботи над тваринами та / або людьми не проводилось.
ORCID
Mohammed A. Abosheasha
https://orcid.org/0000-0002-8866-0040
Afnan H. El-Gowily
https://orcid.org/0000-0001-5290-7278
REFERENCES
Bikadi, Z., & Hazai, E. (2009). Application of the PM6 semi-empirical method to modeling proteins enhances docking accuracy of AutoDock. Journal of Cheminformatics, 1(1), 15. https://doi.org/10.1186/1758-2946-1-15
Chan, J. F. W., Lau, S. K. P., To, K. K. W., Cheng, V. C. C., Woo, P. C. Y., &Yue, K. Y. (2015). Middle East respiratory syndrome coronavirus:Another zoonotic betacoronavirus causing SARS-like disease. Clinical Microbiology Reviews, 28(2), 465–522. https://doi.org/10.1128/CMR.00102-14
Chen, Y., Guo, Y., Pan, Y., & Zhao, Z. J. (2020). Structure analysis of the receptor binding of 2019-nCoV. Biochemical and Biophysical Research Communications, 525(1), 135–140. https://doi.org/10.1016/j.bbrc.2020.02.071
Devaux, C. A., Rolain, J.-M., Colson, P., & Raoult, D. (2020). New insights on the antiviral effects of chloroquine against coronavirus: What to expect for COVID-19? International Journal of Antimicrobial Agents, 55, 105938. https://doi.org/10.1016/j.ijantimicag.2020.105938
Harrison, C. (2020). Coronavirus puts drug repurposing on the fast track. Nature Biotechnology, 38, 379–381. https://doi.org/10.1038/d41587-020-00003-1
Huey, R., Morris, G. M., Olson, A. J., & Goodsell, D. S. (2007). A semiempirical free energy force field with charge-based desolvation. Journal of Computational Chemistry, 28(6), 1145–1152. https://doi.org/10.1002/jcc.20634
Jin, Z., Du, X., Xu, Y., Deng, Y., Liu, M., Zhao, Y., … Yang, H. (2020). Structure of Mpro from COVID-19 virus and discovery of its inhibitors. Nature, 582, 1–9. https://doi.org/10.1038/s41586-020-2223-y
Li, G., Fan, Y., Lai, Y., Han, T., Li, Z., Zhou, P., … Wu, J. (2020). Coronavirus infections and immune responses. Journal of Medical Virology, 92(4), 424–432. https://doi.org/10.1002/jmv.25685
Lu, R., Zhao, X., Li, J., Niu, P., Yang, B., Wu, H., … Tan, W. (2020). Genomic characterisation and epidemiology of 2019 novel coronavirus: Implications for virus origins and receptor binding. The Lancet, 395 (10224), 565–574. https://doi.org/10.1016/S0140-6736(20) 30251-8
Morris, G. M., Goodsell, D. S., Halliday, R. S., Huey, R., Hart, W. E., Belew, R. K., & Olson, A. J. (1998). Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function. Journal of Computational Chemistry, 19(14), 1639–1662. https://doi.org/10.1002/(SICI)1096-987X(19981115)19:14<1639::AID-JCC10>3.0.CO;2-B
NCT04365309. (2020). Protective effect of aspirin on COVID-19 patients. Retrieved from https://clinicaltrials.gov/ct2/show/study/NCT04365309
Obi, A. T., Tignanelli, C. J., Jacobs, B. N., Arya, S., Park, P. K.,
Wakefield, T. W., … Napolitano, L. M. (2019). Empirical systemic anticoagulation is associated with decreased venous thromboembolism in critically ill influenza a H1N1 acute respiratory distress syndrome patients. Journal of Vascular Surgery: Venous and Lymphatic Disorders, 7 (3), 317–324. https://doi.org/10.1016/j.jvsv.2018.08.010
Ou, X., Liu, Y., Lei, X., Li, P., Mi, D., Ren, L., … Qian, Z. (2020). Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV. Nature Communications, 11 (1), 1620. https://doi.org/10.1038/s41467-020-15562-9
Paules, C. I., Marston, H. D., & Fauci, A. S. (2020). Coronavirus infectionsmore than just the common cold. JAMA – Journal of the American Medical Association, 323, 707–708. https://doi.org/10.1001/jama.2020. 0757
Pushpakom, S., Iorio, F., Eyers, P. A., Escott, K. J., Hopper, S., Wells, A., …Pirmohamed, M. (2018). Drug repurposing: Progress, challenges and recommendations. Nature Reviews Drug Discovery, 18, 41–58. https://doi.org/10.1038/nrd.2018.168
Ramphul, K., & Mejias, S. G. (2020). Coronavirus disease: A review of a new threat to public health. Cureus, 12(3), e7276. https://doi.org/10.7759/cureus.7276
Salata, C., Calistri, A., Parolin, C., & Palù, G. (2019). Coronaviruses: A paradigm of new emerging zoonotic diseases. Pathogens and Disease, 77(9), ftaa006. https://doi.org/10.1093/FEMSPD/FTAA006
Salentin, S., Schreiber, S., Haupt, V. J., Adasme, M. F., & Schroeder, M. (2015). PLIP: Fully automated protein-ligand interaction profiler. Nucleic Acids Research, 43(W1), W443–W447. https://doi.org/10.1093/nar/gkv315
Sohag, A., Hannan, M., Rahman, S., Hossain, M., Hasan, M., Khan, M., … Uddin, M. (2020). Revisiting potential druggable targets against SARSCoV-2 and repurposing therapeutics under preclinical study and clinical trials: A comprehensive review. Drug Development Research, 21709. https://doi.org/10.1002/ddr.21709
Solis, F. J., & Wets, R. J. B. (1981). Minimization by random search techniques. Mathematics of Operations Research, 6(1), 19–30. https://doi.org/10.1287/moor.6.1.19
Stewart, J. J. P. (2009). Application of the PM6 method to modeling proteins. Journal of Molecular Modeling, 15(7), 765–805. https://doi.org/10.1007/s00894-008-0420-y
Tremblay, D., van Gerwen, M., Alsen, M., Thibaud, S., Kessler, A. J., Venugopal, S., … Naymagon, L. (2020). Impact of anticoagulation prior to COVID-19 infection: A propensity score-matched cohort study. Blood, 136(1), 144–147. https://doi.org/10.1182/blood.2020006941
Walls, A. C., Park, Y.-J., Tortorici, M. A., Wall, A., Mcguire, A. T., & Correspondence, D. V. (2020). Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell, 181, 281–292. https://doi.org/10.1016/j.cell.2020.02.058
Wang, Z., Chen, X., Lu, Y., Chen, F., & Zhang, W. (2020). Clinical characteristicsand therapeutic procedure for four cases with 2019 novel coronavirus pneumonia receiving combined Chinese and Western medicine treatment. Bioscience Trends, 14(1), 64–68. https://doi.org/10.5582/BST.2020.01030
World Health Organization (WHO). (2020). Coronavirus disease (COVID-2019) situation reports: Situation Report-92. Retrieved from https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports
Xu, X., Chen, P., Wang, J., Feng, J., Zhou, H., Li, X., … Hao, P. (2020). Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission.Science China Life Sciences, 63, 457–460. https://doi.org/10.1007/s11427-020-1637-5
How to cite this article: Abosheasha MA, El-Gowily AH. Superiority of cilostazol among antiplatelet FDA-approved drugs against COVID 19 Mpro and spike protein: Drug repurposing approach. Drug Dev Res. 2020;1–13. https://doi.org/10.1002/ddr.2174
Стаття надана мовою оригіналу





